封面图
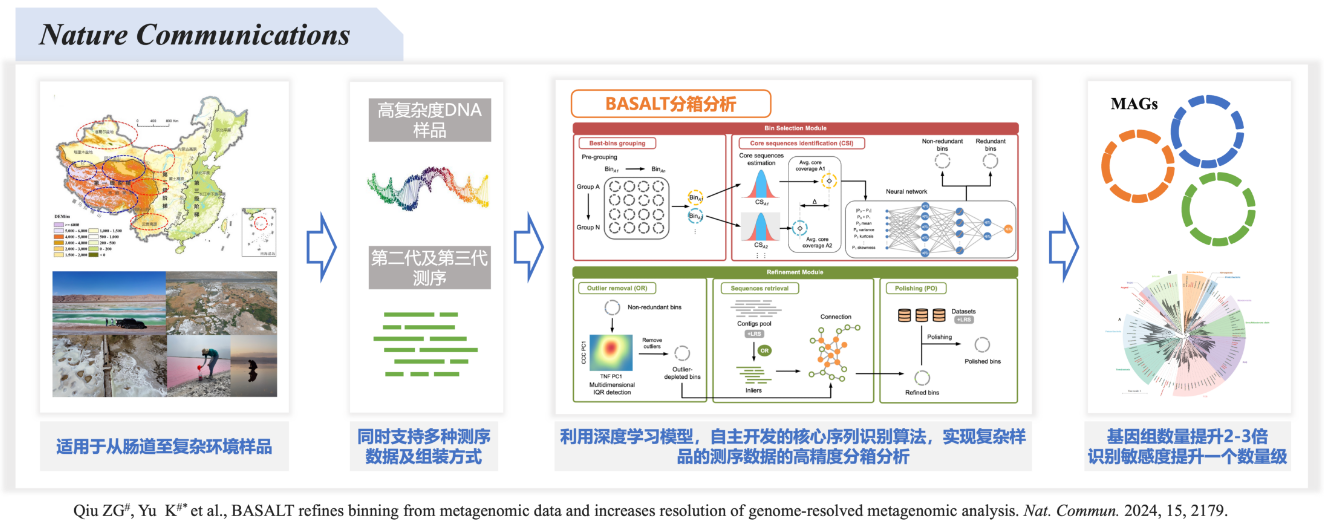
地球上生活着大约800-1000万种微生物。然而,从微生物发现至今的300多年间,人类分离培养的微生物不足2万种,这暗示着未被人类发现或培养的微生物类群占到地球所有微生物类群的99%以上。这些类群主要的来自于各种生境的低丰度(低细胞数)物种,它们被类比为微生物“暗物质”。在近10余年间,高通量测序技术的发展赋予了研究者不依赖于微生物的纯培养,仅通过对环境样品进行直接测序及分析即可获得这些微生物基因组的能力。宏基因组分箱(Binning)方法是实现从测序数据中直接获取微生物基因组最为重要的分析技术之一,其可对微生物“暗物质”及其功能进行资源化,为挖掘未知的微生物功能类群(如肠道疾病治疗、环境修复可用类群)、解析它们具有潜在药用、工业或环境价值的生物合成、降解基因簇(如新型抗生素、抗菌肽、工业酶制剂)提供了重要的方法学基础。然而,现阶段从二代和三代宏基因组测序数据中恢复宏基因组组装的基因组(Metagenome-assembled genomes, MAGs),特别是对于低丰度微生物基因组的恢复效率仍然很低。2024年3月,我院余珂研究团队在Nature Communications (NC)上发表了研究论文“BASALT refines binning from metagenomic data and increases resolution of genome-resolved metagenomic analysis”,该研究中开发的软件BASALT(Binning Across a Series of AssembLies Toolkit)可对二代和三代宏基因组序列进行高效的分箱和优化,并最终实现了对宏基因组测序数据的高效利用,大量获取低丰度微生物的基因组。这一突破性的进展将为我们深入了解微生物世界的奥秘、挖掘其应用潜力提供重要的方法学基础。
BASALT软件运用并整合了多个主流的分箱软件,利用基于深度学习开发的核心序列识别算法找出组装后基因组的核心序列,进行去冗余、去污染、片段找回等一系列基因组优化步骤,可以显著提高MAGs质量和菌株水平的分辨率。此外,BASALT还将三代测序的长片段序列应用至上述一系列优化步骤中,大大提高了三代测序数据的利用效率。以标准数据集进行测试的结果表明,BASALT可获得较其他同类软件(VAMB [Nature Biotechnology, 2021],DAStool [Nature Microbiology, 2018],MetaWRAP [Microbiome, 2018])2倍以上的高质量基因组。更为重要的是,在对盐碱湖表层沉积物的实际样品数据中,BASALT对低丰度基因组的识别灵敏度较其他三个软件提高了一个数量级,这为人类发掘存在于特定环境中的低丰度物种,利用微生物“暗物质”资源,提供了崭新的技术方法。

在相关研究中,余珂团队将BASALT技术应用于多种样品,包括垃圾渗滤液处理场、可高效除氮的藻菌共生体、厌氧氨氧化反应体系、中国西北部盐碱湖泊的微生物组的解析之中,这些研究不仅发现了5000余种新型的微生物物种,还解析了细菌与古菌之间、藻类与细菌之间多种物质交换关系。同时,研究团队也发现了大量的生物合成和降解基因簇。论文分别发表在环境科学与工程的顶级期刊Water Research (Nature index杂志,WR),Environmental Science & Technology (Nature index杂志,ES&T), Environmental International (EI),及Environmental Science and Ecotechnology (ESE)之中。这些研究都印证了BASALT强大的宏基因组分箱能力,为未来进一步发掘这些体系中的微生物“暗物质”的保障奠定了坚实的基础。
以上研究得到了南燕生态环境研究实验室的倪晋仁院士的大力支持,获得了北京大学AI4S团队的田永鸿教授、袁粒教授、陈杰教授在深度学习模型的开发上的强力支持,同时得到了环境与能源学院全方位的支持。深研院接续学者计划特聘副研究员仇知光博士(NC、ESE),在读博士生耿燕妮(ES&T),博士毕业生邓春芳(WR),博士后乔雪姣博士(EI)等为论文的第一作者,张丽娟博士(EI)等为部分论文的共同通讯作者;余珂为以上论文的唯一通讯作者(NC、WR、ESE)或共同通讯作者(ES&T、EI)。这些研究得到了包括中国国家重点研发计划(项目编号:2021YFA1301300)、中国国家自然科学基金(项目编号:51939009)及北京大学科学智能(AI4S)专项等项目的资助。
上述发表的论文列表(#表示第一作者,*表示通讯作者):
(1) Z Qiu#, C Lian, L Yuan, Y Hong, K Yu#* et al., BASALT refines binning from metagenomic data and increases resolution of genome-resolved metagenomic analysis. Nature Communications, 2024, 15, 2179.
(2) C Deng#, T Chen#, Z Qiu, H Zhou, B Li, Y Zhang, X Xu, CA Lian, X Qiao, K Yu*. A mixed blessing of influent leachate microbes in downstream biotreatment systems of a full-scale landfill leachate treatment plant. Water Research, 2024, 253: 121310.
(3) Y Geng#, Z Xiong, L Yang*, CA Lian, S Pavlostathis, Z Qiu, H Chen, Q Luo, Y Liu, Z Liu, P Shao, J Zou, H Jiang, S Luo, K Yu*, X Luo*, Bidirectional enhancement of nitrogen removal by indigenous synergetic microalgal–bacterial consortia in harsh low-C/N wastewater. Environmental Science & Technology, 2024.
(4) X Qiao, L Ding, F Fang, C Fu, R Wei, Y Chen, S Zheng, X Wang, Y Yan, K Yang, N Xu, H Tao, K Yu*, L Zhang*, An integrated meta-omics approach reveals the different response mechanisms of two anammox bacteria towards fluoroquinolone antibiotics, Environmental International, 2024, 185: 108505.
(5) Z Qiu#, Y Zhu, Q Zhang, X Qiao, R Mu, Z Xu, Y Yan, F Wang, T Zhang, K Yu*. Unravelling Biosynthesis and Biodegradation Potentials of Microbial Dark Matters in Hypersaline Lakes. Environmental Science and Ecotechnology, 2024, 20, 100359.